BioPerl でTaxonomy IDだの生物種名だの
BioSampleっていうデータベースができまして、実験サンプルの情報が収載されるようになったわけですが、今までTaxnomy IDで管理されていたのがBioSample IDになってTaxonomy IDが振られなくなったなどと騒いでいるのが世に言うTaxonomy ID問題ってやつだったりします。
そんな中、自分のやっておるDBCLS SRA ( http://sra.dbcls.jp/ ) では、前のバージョンで(って現バージョンにがっちゃんこできていないってやつですが)生物種からの検索機能を強化していまして、イネで調べたらその下のindicaやjaponicaも出るようにするなどという機能をつけております。
最近、どうもそのデータベースの更新がコケていたようで、それはそれでご迷惑をおかけしとったわけですが、さらによく調べると、この機能が重い重い。全然 反応が返ってこない。いろいろと調べた結果、IDと名前の対応、親子関係を抽出するのに、ローカルのファイルを見ていたところであったことがわかりました。具体的には
my $dbh = Bio::DB::Taxonomy->new( -source => 'flatfile',
-directory => '/tmp/',
-nodesfile => '/share/data/taxonomy/nodes.dmp',
-namesfile => '/share/data/taxonomy/names.dmp');
これをやると、nodes.dmp と names.dmp から/tmpにインデックスファイルを作成して、それをみてうねうねする、というような仕様のようで、で、処理の度にインデックスファイルを作成→終わったら消すをやっていた模様。どうせなら、そのインデックスファイルをそのままにしておいて、そこ指定するとひっぱってこれる、というような仕様にするとかすればよいのに。
ちなみに、こうやって読み込んだ後の処理の例として
$taxon_in_obj = $dbh->get_taxon(-name => $taxon_in);
if ($taxon_in_obj ne "") {
$taxon_id = $taxon_in_obj->id;
}
生物種名を入れて、Taxonomy IDを出すあたりがこれです(本当はもっと短く書ける)。
で、つどつどインデックスファイル作って消して、が時間がかかるようなので、つどつどNCBIに訊くに変更。
my $dbh = Bio::DB::Taxonomy->new( -source => 'entrez');
それだけで、読み込んだ後の処理の部分はまったくいじらずで大丈夫。
これで数十秒(場合によっては分単位)かかっていたのが、数秒で結果で返ってくるようになりましたとさ。
STAP細胞関連のNGSデータを論文から読み取ってアクセスしやすくしてみた
3行まとめ
- http://trace.ddbj.nig.ac.jp/DRASearch/study?acc=SRP038104 にそろっている
- 今回のは、ID記載のマジックとNCBIのインターフェースのラビリンス具合の合わせ技
- わしわし登録するところは研究所内に登録アシストの人を置いたら? そうでなくても、とりあえずDDBJに相談しなよ
どうも、公共NGSデータをアクセスしやすくしている者です。
一応、ベタに背景を述べるとですね。NGSデータはDDBJ/EBI/NCBI (アルファベット順)の3つのセンターに登録窓口があって、どこに登録されてもいずれはデータ交換されるからどこに登録してもいいんですけど、SRA (Sequence Read Archive) (広義。DDBJのとEBIのとNCBIのを合わせた名称)として公開されて、誰でも利用できるようになっております。で、うちらはDDBJと協力をして、この登録データをいろんな観点から+簡単に見つけられるようにしよう、ってことをやっていて、DBCLS SRA ( http://sra.dbcls.jp/ ) ってのを作って公開しております。
このいろんな観点から、というのは、目的別(ゲノムか、発現か、エピゲか)、機器別(MiSeq とか HiSeq とか PacBio とか)、生物種別とかで、加えて、論文からというのをやっております ( http://sra.dbcls.jp/cgi-bin/publication.cgi ) 。今回みたいに、この論文のデータがほしいな、とかいうときに便利なんでね。
さっそく、実際の記述を見てみると、こんな感じになっておりました。
Bidirectional developmental potential in reprogrammed cells with acquired pluripotency
http://www.nature.com/nature/journal/v505/n7485/full/nature12969.html
RNA-seq and ChIP-seq files have been submitted to the NCBI BioSample databases under accessions SAMN02393426, SAMN02393427, SAMN02393428, SAMN02393429, SAMN02393430, SAMN02393431, SAMN02393432, SAMN02393433, SAMN02393434 and SAMN02393435.
オープンアクセスになったので、こういうときに便利ですゎね。
しかし、ここに出てくるのはSRAのIDではないんですよね。。。まぁ、順々に。
ここで皆様にまず訴えたいのはですね、SRAに登録されたデータの構造ですね。そこは本家DDBJの図がよくできているので、それを引用。
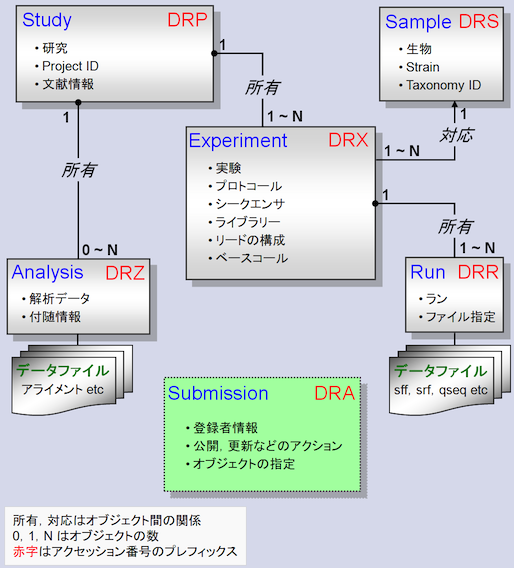
実際のシーケンスデータに加えて、メタデータとして実験情報の書かれたファイルがあってですね、そこに記載の情報を元手に検索をかけるわけですが、以下の感じになっております
- study:プロジェクト情報(例:1000人日本人ゲノム読みますプロジェクト)
- experiment:実験情報。使用機器とか試薬の条件とか(例:1000人読む各々の個人に対しての測定)
- sample:実験サンプル情報(例:1000人各々の個人についての情報。性別とか年齢とか)
- run:実際に読んだシーケンスについての情報
- analysis:解析した情報(optional)
- submission:上のをまとめて登録した際の登録そのものに対しての情報(てかIDとか登録日とか)
で、各々、SRP000001とかSRXとかそういうIDが振られていて、それで論文に引用すればいいわけです。
All Solexa reads have been deposited in the Read Archive at DDBJ http://www.ddbj.nig.ac.jp/ with accession number DRA000001, ...
ちょっと凝るとこんなのもありますわな。
we chose to analyse the largest samples: ERR011058 (10BF), ERR011062 (12BF) and ERR011080 (5EU) (21811, 25724 and 22714 sequences, respectively), ...
こういうのも内部ではヨサゲに処理しております。
で、見ていると、こんなのも出てきます。
All the DNA sequencing data in this work is deposited at GEO with an accession number of GSE15188.
一見、マイクロアレイっぽいんですが、DNA sequencing とあるのでNGSデータであります。とある事情*1により、発現データは、GEOに(も)登録されるようになっております。実際には、発現関連のNGSデータはまずGEO(とかEBIだとArrayExpress)に登録されて、内部(NCBIとかEBI)でそのデータがSRAに送られてそちらでも公開、という流れですわな。
すると、同じデータがGEOとSRAに登録されるという状況があるわけです(他に、NGSデータはSRAに、それをつないだゲノムデータはGenBank/EMBL/DDBJ に登録ということもある)。とすると、そいつを束ねるデータベースがいりそうですよね、ということで、プロジェクトのデータベースとサンプルのデータベースができて、各々が BioProject と BioSample というデータベースになっております。図式化すると、以下のような感じ。
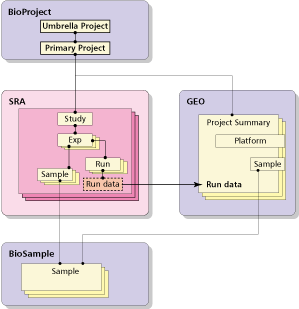
ゆくゆくはSRAなら、StudyとSampleの部分が、BioProjectとBioSampleに置き換えられる予定(だったはず)。
さてさてお待たせしました。で、件の記述はこのBioSampleのIDでの記述だったわけです。
で、記述にあるIDをBioSampleのページ( http://www.ncbi.nlm.nih.gov/biosample )で検索すると*2、 http://www.ncbi.nlm.nih.gov/biosample/?term=SAMN02393426 な感じになりまして、これを見ると、下にBioProjectへのリンクが。で、これをたどると、真ん中の表に SRA Experiments: 42とあるので42コあるんだな、と。この42をクリックすると、NCBIのSRAのサイトに飛んで、確かに42コのSRA Experiment IDが並んでいるわけですが、これをぽちぽちたどってデータを落とすか??? ということになったわけですよ。で、先ほど訴えたSRAのデータ構造に戻るわけですが、Experimentをまとめる形でStudyとかSubmissionのIDがあるはずだと。NCBI のSRAのサイトはラビリンスですから、入らないのが懸命です。さっそく、DDBJのサイト( http://trace.ddbj.nig.ac.jp/DRASearch/ )で検索しましょう。とりあえず、BioSample → BioProject → SRA Experiment ときてますが、一番上のIDで検索して http://trace.ddbj.nig.ac.jp/DRASearch/experiment?acc=SRX472668 が得られるわけです。すると右の表にStudy IDはSRP038104だとあるので、これをぽちっとして http://trace.ddbj.nig.ac.jp/DRASearch/study?acc=SRP038104 でゴールです。(全部のExperimentがこのStudyにぶら下がることを確認済)。右の表からわしわし落として、遊びましょう。
ちなみに、作っているDBCLS SRA ( http://sra.dbcls.jp/ )には、Hot Publicationsという欄が一番下にあって、そこからもリンクしてあります。
*1:通称:NCBI SRAやめます事件。オバマ大統領が予算カットだとか言ったあおり(←ちょっと話が歪んどるな)http://www.nlm.nih.gov/pubs/techbull/jf11/jf11_ncbi_reprint_sra.html とか
*2:DDBJにもBioSampleがありますが、現時点で検索できるのはDDBJに登録されたもののみ
名古屋議定書に関するパブコメ明日まで。
適当にとりあえずメモっとくわ。
名古屋議定書のって、ある国から何か(植物でも昆虫でも)もってきた → 薬作った(そして儲けた)→ 作った方の国だけ儲けて不公平でね? という背景だと記憶しているので、生き死には関係ないのではないか( https://twitter.com/chalkless/status/426275932815364096 )
平成25年12月27日
「名古屋議定書に係る国内措置のあり方検討会報告書(案)」 に対する意見の募集(パブリックコメント)について (お知らせ) http://www.env.go.jp/press/press.php?serial=17565
dantyuteiさんによるよくわかる解説:http://d.hatena.ne.jp/dantyutei/20140121
とりあえず書いた理由(こんなに長くなくていいと思う。あと、あっているかどうか)
名古屋議定書は、遺伝子資源利用国が、その遺伝子資源を活用し、
過剰な利益を得ることによって、本来、遺伝子資源提供国が得られる利益が
著しく減ることによる不公平を是正するためのものであると理解している。
我が国は、人々の動植物への関心が非常に高く、端的には、一般の書店で
花、草木、魚、昆虫、小動物といった幅広い動植物に渡る大小の図鑑が
容易に手に入るが、欧米をはじめとした諸外国では、このような状況に
ないのが現状である。
このことは、これらの動植物を趣味で収集、飼育栽培する一般の人々が
多いという環境を生んでおり、また、趣味を究めた在野の研究者が多い
というのも我が国の特徴と言えよう。
在野の研究者の例では、日本では蝶の生態(食草、産卵や幼虫期の時期、
越冬態等)が非常に詳細に明らかにされているが、これらは、もともと
一般の人々が趣味で観察、飼育をし、明らかにしたものである
(著名な人としては五十嵐邁 氏が挙げられよう)。
昆虫を例にとれば、定期的に標本や生体を交換・売買する
インセクトフェアといった催しが行われ、また、むし社をはじめ、
店舗で昆虫を購入することにより、自宅等での飼育、観察を行う環境が
現状ある。(そしてたとえば飼育のノウハウはブログや書籍にまとめられ
誰もが情報を得られるようになっている)
名古屋議定書の趣旨からすると、このような個人の趣味や在野の研究は
遺伝子資源を活用する範疇からは明らかに外れており、
遺伝子資源提供国の利益を妨げるものではない。
逆に、工芸品としてアジアを中心に売られている蝶やコガネムシの
標本の購入ができなくなると思われ、手続きを要求する方が利益を妨げると思われる。
よって、これら個人の趣味や在野の研究、あるいは、それらを対象にした
標本商や零細輸入業者に関しては、手続きの免除、あるいは大幅な簡素化が
適当であるという結論に至るものである。
→実際に報告書のどこにツッコミを入れるんだよ、というところはまだ見きれていない。
Mac miniにSSDを増設してみた(落ち穂拾い)
落ち穂拾いという言葉は、意外と便利な言葉な気がします。差分だけ書いて落ち穂拾いって言えばいいんですからね。まぁ、そんなことはどうでもいいのですが、個人的には垣間見るも便利な言葉だと思っています。はい、どうでもいいです。
えっと、3月だかにMac miniを購入しました。いつもはメモリにディスク増し増しで注文なのですが、今回は自宅用ですし、そこはシンプルプランにして、秋葉原でメモリとSSDを購入し、自分でトッピングしました(トッピングいうな)。
実際の増設は[Mac] Mac mini 2012のHDDを「SSD」に換装して快適Macライフを手に入れる! | Tools 4 Hackあたりとか、まぁ、YouTube探せば動画もあると思いますが、いくつかのを参考にしながらやっていたら、各々で順番が違うので、一つ、コネクタを外し忘れ、断線させました。。。電源ランプのLED用だったので事なきを得ていますが、あぶないあぶない。。。メモリの増設はやさしいですが、ディスク換装はけっこう覚悟がいるなぁ、などと思いました。。。
で、今回は換装でなくて増設していて、元ある1TBのHDDに、250GBのSSDという構成になっております。システムがSSDで、データがHDDね。
Macでは、こうやってSSD+HDDの構成だと、あたかも1つのドライブであるかのようにするFusion Driveのにできたりします。が、今回はFusion Driveにしてません。
その後、Command + R で電源ON(これでダメなら オプションキーを押しながらONかな)。すると、いろいろメニューが出てきたりするわけです。で、SSDのフォーマットをしよう、などと書いてあったりするわけですが、今回はFusion Driveにしないので、ディスクユーティリティを立ち上げてはいけません。なんかエラーっぽいメッセージが出てですね、うっかりOKしたりすると、無理矢理 Fusion Driveにさせられるのです。。。(しかも数時間の時間がかかる)。そこをすっ飛ばして、いきなりOSのインストールです。申し遅れましたが、今回は10.8のMountain Lionなのでネット経由でのインストールなのであります(そもそもCDとかDVDを入れるスロットがない)。再インストールを選択すると名称未設定みたいな名前のディスクが2つ並んでどっちかなーみたくなるのですが、そこは容量でわかるでしょ。
もし仮にFusion Driveになってしまったら、ディスクユーティリティとかOS再インストールとか出るメニューで上の文字メニュー(一般的にファイルとか編集とか書いてあるところ)のユーティリティあたりを開くとターミナルがあるのでそれを開いてコマンドで設定解除です。具体的には[Mac] 噂のFusion Driveを自分のSSD&外付けHDDを使って自家製で構築してみる。 | Tools 4 Hackに書いてあります(ちなみにこの記事の本題はFusion Driveにする方で逆操作なのですが)。
で、無事にOSが起動されるわけです。
で、ここからまた設定なわけですね。
まずは、SSDの設定でTRIMの有効化ってのがあるらしい。これは最初の記事に載ってます。TRIM Enablerってのを落としてきて有効にするだけ。もしくは、ドメインパーキングを参考に。
で、その次で、この記事にも載っているのですが、ホームディレクトリをHDDの方にすること。システム環境設定 > ユーザとグループ → ユーザアイコンを右クリック → 詳細オプション → ホームディレクトリを選択。な感じらしい。
ここが原因かわからんのですが、QuickTimeがおかしくてですね、終了できなかったり、ウインドウが消えなかったり、ボタンが表示されなかったり、いろいろ起きて結局 再インストールになったのですが、もうちょっとエラーを探ってみたいとは思っとります。
というのも、10.7から ~/Library はFinderから非表示になっているようで、うっかりここを壊したんじゃないか、もしくはPermissionが変なのではないか、などと思っているからなのですが。常に表示させたいので非表示のユーザーライブラリフォルダーにアクセスする方法(Mac OS X 10.7 以降)を参考にすることに。
あとは何だ。Web共有か。前は共有でWeb共有とすると~/Sites/以下が公開されていたけれども、そのメニューがなくなったと。OS X 10.8 Mountain Lion 向け、Web共有を手動で有効にする方法 | Macとかの雑記帳を見て設定(再起動しても自動であがるんだっけか?)
とりあえず、こんなもんかな。また書くことがあれば追記するってことで。
Google Chart のグラフをベクターで扱う(そんなんかよ)
ぶはは。ここに書くのも1年ぶりかよ。いったい今まで何やってたんだよ感も漂いますが、まぁいろいろやってましたってことで(だから何を)
えーっと、昨日、論文のreiviseを投稿しました。
reviewerのコメントの中に、図が汚いというのがあって、まぁ、これは新規投稿の際にわかっていたのですが、なぜか図のquality checkプログラムを通ってしまったため(って、人の目ににじんだ画像は機械ににじんでいるとはわからない気もするわけで)、ほったらかしていたわけです。
てなわけで、こんなグラフを載せております。
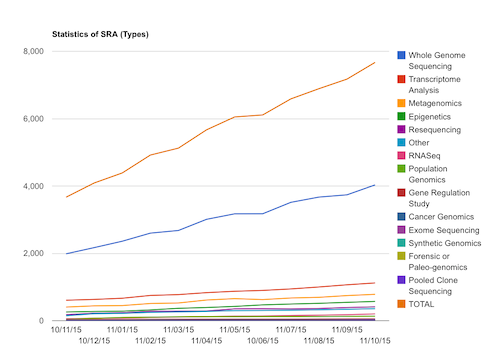
もともとは、 http://sra.dbcls.jp/sra.stat.html あたりに載せているグラフなわけですが、Google Chart を利用しております。が、このグラフ、SVG で出力されているにもかからわず、右クリックで(PNGでも)保存とかできないんですよね。
なら、Google ドキュメント(現:Google Drive)で数値を入れて、で、それを保存しようってぇと、http://support.google.com/drive/bin/answer.py?hl=ja&answer=63835 PNG保存はできるっぽい。しかし、グラフに補足でラベルをつけたいし、どうにかベクター画像にしてイラレで編集したいなーと。
いろいろググったけれども、よさげなサイトは出ず。。。
で、もうメンドイから、ダメもとで、元のサイトで印刷とやってPDF保存(こちとらMac + Chrome なので印刷ダイアログのPDFをプレビュー)とやって、それをイラレで開いてみた。らば、ちゃんと折れ線グラフがベクター化されて、太さとか選べるし。文字はアウトライン化されてしまったので、ラベルが長すぎて ... とかにされたところを直すために打ち直したけれども、そのままくいっと拡大・縮小できるし、よかったよかった。
文献リストの手直しメモ
毎度おなじみながら、NGSの公共データベースであるSRAで、メタデータのまとめをして統計的なところから検索するっていう SRAs ってのをやってます ( http://sra.dbcls.jp/ )。この中で、文献からデータを検索するってのがあるわけですが、5月下旬にNGS現場の会 第2回研究会ってのに参加しまして、いろんなベンダーあるなーとか思ったわけです。んでもって、(あくまでこちらでつかんでいる情報でだけですが)文献が出ているデータを機種別に検索してみまして
- +
platform count(*)
- +
Illumina Genome Analyzer II 4298 454 GS FLX 3701 Illumina Genome Analyzer 2927 454 GS FLX Titanium 847 Illumina Genome Analyzer IIx 645 Illumina HiSeq 2000 377 AB SOLiD System 3.0 154 454 GS 20 154 Helicos HeliScope 115 AB SOLiD System 2.0 114 AB SOLiD System 66 AB SOLiD 4 System 56 454 GS 21 NULL 18 unspecified 15 PacBio RS 13 Complete Genomics 4 454 GS Junior 2 Ion Torrent PGM 2 Illumina MiSeq 1
- +
こんな結果を得ました。
で、ほほぅ。Illumina MiSeqもあるのかー、とMySQLをたたいたらば
- +
RA PLATFORM pmid
- +
SRA048303 Illumina MiSeq 22185227
- +
確かにあったわけです。
ところが、実際にサービスしているページ ( http://sra.dbcls.jp/cgi-bin/publication.cgi ) では出ない。これはおかしい。
もうちょっと調べてみますと
- +
RA study_title platform
- +
SRA048303 Ultra-Deep Targeted Sequencing Illumina Genome Analyzer IIx SRA048303 Ultra-Deep Targeted Sequencing Illumina MiSeq
- +
と、1つの登録で複数のプラットフォームであったわけです。なるほどなるほど。
で、どこでコケるのかなーっと思ったらば、取ってきた文献とデータのペアをMySQLに登録するプロセスで
if len(rows) != 1: # データは一種類だけ入っているはずなので、複数あると
おかしい
とある行を発見したのですが、データは一種類ではなかった。。。(残念)
というわけで、今度 直してねー(と、とりあえず書いておく)