STAP細胞関連のNGSデータを論文から読み取ってアクセスしやすくしてみた
3行まとめ
- http://trace.ddbj.nig.ac.jp/DRASearch/study?acc=SRP038104 にそろっている
- 今回のは、ID記載のマジックとNCBIのインターフェースのラビリンス具合の合わせ技
- わしわし登録するところは研究所内に登録アシストの人を置いたら? そうでなくても、とりあえずDDBJに相談しなよ
どうも、公共NGSデータをアクセスしやすくしている者です。
一応、ベタに背景を述べるとですね。NGSデータはDDBJ/EBI/NCBI (アルファベット順)の3つのセンターに登録窓口があって、どこに登録されてもいずれはデータ交換されるからどこに登録してもいいんですけど、SRA (Sequence Read Archive) (広義。DDBJのとEBIのとNCBIのを合わせた名称)として公開されて、誰でも利用できるようになっております。で、うちらはDDBJと協力をして、この登録データをいろんな観点から+簡単に見つけられるようにしよう、ってことをやっていて、DBCLS SRA ( http://sra.dbcls.jp/ ) ってのを作って公開しております。
このいろんな観点から、というのは、目的別(ゲノムか、発現か、エピゲか)、機器別(MiSeq とか HiSeq とか PacBio とか)、生物種別とかで、加えて、論文からというのをやっております ( http://sra.dbcls.jp/cgi-bin/publication.cgi ) 。今回みたいに、この論文のデータがほしいな、とかいうときに便利なんでね。
さっそく、実際の記述を見てみると、こんな感じになっておりました。
Bidirectional developmental potential in reprogrammed cells with acquired pluripotency
http://www.nature.com/nature/journal/v505/n7485/full/nature12969.html
RNA-seq and ChIP-seq files have been submitted to the NCBI BioSample databases under accessions SAMN02393426, SAMN02393427, SAMN02393428, SAMN02393429, SAMN02393430, SAMN02393431, SAMN02393432, SAMN02393433, SAMN02393434 and SAMN02393435.
オープンアクセスになったので、こういうときに便利ですゎね。
しかし、ここに出てくるのはSRAのIDではないんですよね。。。まぁ、順々に。
ここで皆様にまず訴えたいのはですね、SRAに登録されたデータの構造ですね。そこは本家DDBJの図がよくできているので、それを引用。
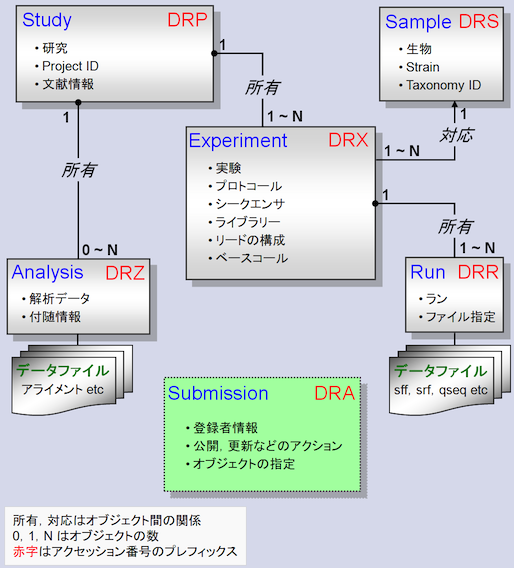
実際のシーケンスデータに加えて、メタデータとして実験情報の書かれたファイルがあってですね、そこに記載の情報を元手に検索をかけるわけですが、以下の感じになっております
- study:プロジェクト情報(例:1000人日本人ゲノム読みますプロジェクト)
- experiment:実験情報。使用機器とか試薬の条件とか(例:1000人読む各々の個人に対しての測定)
- sample:実験サンプル情報(例:1000人各々の個人についての情報。性別とか年齢とか)
- run:実際に読んだシーケンスについての情報
- analysis:解析した情報(optional)
- submission:上のをまとめて登録した際の登録そのものに対しての情報(てかIDとか登録日とか)
で、各々、SRP000001とかSRXとかそういうIDが振られていて、それで論文に引用すればいいわけです。
All Solexa reads have been deposited in the Read Archive at DDBJ http://www.ddbj.nig.ac.jp/ with accession number DRA000001, ...
ちょっと凝るとこんなのもありますわな。
we chose to analyse the largest samples: ERR011058 (10BF), ERR011062 (12BF) and ERR011080 (5EU) (21811, 25724 and 22714 sequences, respectively), ...
こういうのも内部ではヨサゲに処理しております。
で、見ていると、こんなのも出てきます。
All the DNA sequencing data in this work is deposited at GEO with an accession number of GSE15188.
一見、マイクロアレイっぽいんですが、DNA sequencing とあるのでNGSデータであります。とある事情*1により、発現データは、GEOに(も)登録されるようになっております。実際には、発現関連のNGSデータはまずGEO(とかEBIだとArrayExpress)に登録されて、内部(NCBIとかEBI)でそのデータがSRAに送られてそちらでも公開、という流れですわな。
すると、同じデータがGEOとSRAに登録されるという状況があるわけです(他に、NGSデータはSRAに、それをつないだゲノムデータはGenBank/EMBL/DDBJ に登録ということもある)。とすると、そいつを束ねるデータベースがいりそうですよね、ということで、プロジェクトのデータベースとサンプルのデータベースができて、各々が BioProject と BioSample というデータベースになっております。図式化すると、以下のような感じ。
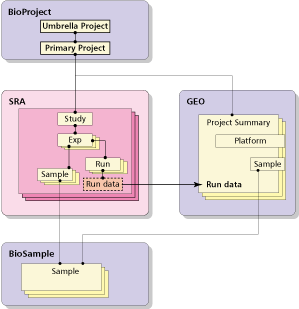
ゆくゆくはSRAなら、StudyとSampleの部分が、BioProjectとBioSampleに置き換えられる予定(だったはず)。
さてさてお待たせしました。で、件の記述はこのBioSampleのIDでの記述だったわけです。
で、記述にあるIDをBioSampleのページ( http://www.ncbi.nlm.nih.gov/biosample )で検索すると*2、 http://www.ncbi.nlm.nih.gov/biosample/?term=SAMN02393426 な感じになりまして、これを見ると、下にBioProjectへのリンクが。で、これをたどると、真ん中の表に SRA Experiments: 42とあるので42コあるんだな、と。この42をクリックすると、NCBIのSRAのサイトに飛んで、確かに42コのSRA Experiment IDが並んでいるわけですが、これをぽちぽちたどってデータを落とすか??? ということになったわけですよ。で、先ほど訴えたSRAのデータ構造に戻るわけですが、Experimentをまとめる形でStudyとかSubmissionのIDがあるはずだと。NCBI のSRAのサイトはラビリンスですから、入らないのが懸命です。さっそく、DDBJのサイト( http://trace.ddbj.nig.ac.jp/DRASearch/ )で検索しましょう。とりあえず、BioSample → BioProject → SRA Experiment ときてますが、一番上のIDで検索して http://trace.ddbj.nig.ac.jp/DRASearch/experiment?acc=SRX472668 が得られるわけです。すると右の表にStudy IDはSRP038104だとあるので、これをぽちっとして http://trace.ddbj.nig.ac.jp/DRASearch/study?acc=SRP038104 でゴールです。(全部のExperimentがこのStudyにぶら下がることを確認済)。右の表からわしわし落として、遊びましょう。
ちなみに、作っているDBCLS SRA ( http://sra.dbcls.jp/ )には、Hot Publicationsという欄が一番下にあって、そこからもリンクしてあります。
*1:通称:NCBI SRAやめます事件。オバマ大統領が予算カットだとか言ったあおり(←ちょっと話が歪んどるな)http://www.nlm.nih.gov/pubs/techbull/jf11/jf11_ncbi_reprint_sra.html とか
*2:DDBJにもBioSampleがありますが、現時点で検索できるのはDDBJに登録されたもののみ